Caractérisation de la biodiversité par l’ADNe (Pôle Écla)
Contexte général
La surveillance biologique des écosystèmes aquatiques de surface et notamment des plans d’eau est utilisée dans un cadre réglementaire (directive cadre européenne sur l’eau, DCE) mais également dans le cadre de suivis régionaux ou locaux (e.g. Observatoire des Lacs Alpins, OLA).
Cette surveillance basée sur la biodiversité est réalisée soit au travers d’outils standardisés (e.g. Indice Phytoplancton Lacs, IPLac), soit au travers de suivis de biodiversité de groupes taxonomiques adaptés aux besoins de surveillance spécifiques. Elle permet d’évaluer l’état écologique des masses d’eau, de suivre les effets d’actions de restauration, de détecter et de suivre la dynamique d’espèces nouvellement introduites et leur conséquence sur la faune existante et plus généralement cette surveillance est nécessaire pour faire progresser la connaissance sur le fonctionnement des écosystèmes lacustres et leur trajectoire temporelle.
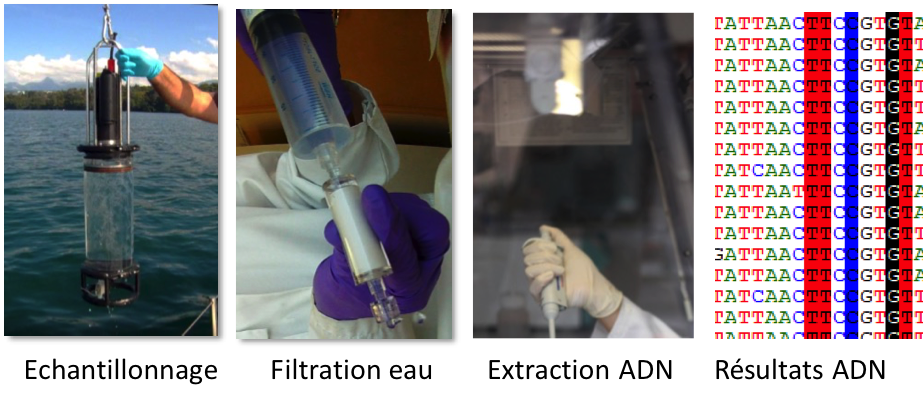
Les méthodes d’échantillonnages varient en fonction des groupes biologiques et l’identification taxonomique est basée sur la reconnaissance morphologique des individus échantillonnés. Ces approches présentent différentes limites selon les groupes biologiques. Pour les communautés piscicoles lentiques, l’échantillonnage (e.g. pose de filets maillants) long et coûteux s’accompagne d’un coût biologique (perte des individus échantillonnés). Pour les microorganismes (phytoplancton, phytobenthos), c’est l’identification taxonomique en microscopie requérant une expertise taxonomique forte qui s’avère limitante (et n’est pas toujours résolutive pour certains groupes comme les bactéries).
Les innovations technologiques en biologie moléculaire (ex. séquençage ADN haut-débit, HTS) permettent d’envisager l’utilisation de l’ADN environnemental (ADNe) comme alternative aux méthodes traditionnelles d’échantillonnage et d’identification. En effet, les organismes peuvent être identifiés par l’échantillonnage de leur ADN intra- ou extra-cellulaire (libre) et le séquençage d’un gène ou fragment de gêne permettant leur reconnaissance. L’exploration de la biodiversité via l’ADNe présente le potentiel d’un échantillonnage non invasif et simplifié (e.g. poissons) et/ou d’une reconnaissance rapide et fiable (e.g. microorganismes). Cette technologie est donc très prometteuse tant pour la biosurveillance que pour l’étude du fonctionnement des écosystèmes lentiques.
Depuis 10 ans, l’INRA et l’AFB contribuent scientifiquement au développement de ces approches sur les compartiments phytobenthos, phytoplancton et poissons. Cette thématique est désormais dynamisée au niveau européen par le biais du réseau DNAqua-Net (COST CA15219, co-animé par l’INRA) et des projets InterReg en cours sur les plans d’eau (SYNAQUA - InterReg France-Suisse co-porté par INRA, Eco-AlpsWater – InterReg Alpine Space avec AFB et INRA comme partenaires français). En ce qui concerne la standardisation, sous l’impulsion du réseau DNAqua-Net, la création d’un nouveau groupe dédié à la standardisation des approches moléculaires pour la biosurveillance des milieux aquatiques vient d’être actée au Comité Européen de Normalisation (CEN). Ces approches nécessitent encore différents développements méthodologiques ainsi qu’un déploiement à large échelle, avant de pouvoir envisager leur standardisation et une utilisation opérationnelle pour une meilleure connaissance et un suivi plus efficace des écosystèmes lacustres. C’est l’objet du présent projet qui focalisera sur le phytobenthos, le phytoplancton et les poissons des plans d’eau nationaux et d’outremer (Petit Saut en Guyane).
Enjeux opérationnels
Cette action recouvre donc à la fois des enjeux méthodologiques et scientifiques :
- Acquérir une meilleure connaissance des potentialités de cette approche basée sur l’ADNe pour caractériser les compartiments biotiques des écosystèmes lacustres ; en particulier ses capacités de quantification (obtenir des inventaires quantifiés), de représentativité spatiale et temporelle.
- Harmoniser et standardiser les approches pour chacun des compartiments biotiques, de l’échantillonnage à l’inventaire de biodiversité, en passant par les étapes de biologie moléculaire, de séquençage et de bioinformatique. De nombreuses étapes restent à préciser, mais il s’agit ici de s’appuyer sur de nombreux résultats déjà acquis par les équipes et de s’inscrire dans une dynamique européenne pour contribuer à l’élaboration de standards,
- Améliorer la caractérisation et la compréhension de la biodiversité des compartiments biologiques de ces écosystèmes et de leurs dynamiques, afin de mieux préserver/restaurer ses écosystèmes souvent emblématiques. Cet enjeu sera abordé grâce aux connaissances des équipes en limnologie et en en coordination avec d’autres actions du pôle.
Tous ces enjeux bénéficieront des expertises scientifiques et techniques complémentaires des équipes du pôle, et de leur capacité à mener conjointement des campagnes d’échantillonnage (vaste réseau, chroniques temporelles, etc).
Publications et rapports
- Valentin V, Frédéric R, Isabelle D, Olivier M, Yorick R, Agnès B (2019) Assessing pollution of aquatic environments with diatoms’ DNA metabarcoding: experience and developments from France Water Framework Directive networks. Metabarcoding and Metagenomics 3: e39646. https://doi.org/10.3897/mbmg.3.39646
(dernière mise à jour : janvier 2020)
A lire aussi : rubrique outils
L’ADN environnemental : une technique innovante pour l’étude de la biodiversité | Page éditoriale
L’ADN environnemental (ADNe) est une technique de surveillance de la biodiversité non invasive pour les milieux naturels et les espèces. Cette approche basée sur la récolte de d'échantillons du milieu (eau, sol, fèces...) permet d’identifier les différents êtres vivants qui résident ou ont traversé ce milieu naturel, y compris des espèces rares.